COOTを使ってみよう
モデル構築界のスター、COOTを使いこなします。
COOTは使いやすいユーザーインタフェースと頻繁なバージョンアップにより、すばらしく高機能なモデリングプログラムになって来ました。基本的な使い方は使ってみればわかる!というぐらい簡単なのでここでは使いこなすための解説をしていきたいと思います。
なお、バージョン間での差異が大きいのでなるべく最新版を使うようにしましょう。最新版はCOOTのページからダウンロードします。
CCP4 6.1.0をインストールしたときに一緒に導入されるのは0.5ですが、私が使っているのは0.5.2です。なお、0.5.0と0.5.2の間には使える機能にかなりの差がありますのでできる限り0.5.2以上を使うほうがいいでしょう。また、バグフィックス等が行われているので可能なら0.6pre1 build 1716以降を使うことをお勧めします。
この手順内でできないことがあれば新しいバージョンを検討して下さい。あくまで私が普段使っている範囲での解説なので詳しい解説はCOOTサイトのドキュメントを読むのが一番いいと思います ![[huh]](image/face/huh.png)
- 01 COOTで行こう! -- 2008-09-11 (木) 17:11:38
- 02 基本のキ -- 2011-05-18 (水) 09:49:36
- 03 さらに使いこなせ! -- 2010-01-06 (水) 10:40:27
- 04 まだまだ便利に -- 2009-05-27 (水) 15:43:22
- 11 スクリプトでハッピー -- 2009-03-02 (月) 17:20:26
- 12 Extensionsを使え! -- 2009-05-19 (火) 11:42:00
- クックブック -- 2012-01-23 (月) 18:02:15
草稿(未分類)
3文字コードがない!分子を扱う -- 0.4.1
PDBに登録されていない化合物を扱うことは多いと思いますが、それを扱うための手順をまとめておきます。
当然、その化合物の座標ファイルがあるという前提です。COOTで分子を扱うためには結合情報およびRestrain情報が必要になります。これはCIFファイルによって提供されています。ここではPRODRGサービスまたはMAKECIFプログラムを使用してCIFファイルを作成する手順を説明します。
1. PRODRGでCIFファイルを作成する
COOTで化合物に対してRegularizeやEdit chi anglesを実行しようとするとその化合物の結合情報などが定義されたCIFファイルが必要となります。新しいバージョンではTorsion generalコマンドが追加されていますが、ちょっと使いにくいです。
そこで、PRODRGサーバを使って座標ファイルからCIFファイルを作成します。
まずPRODRGサーバのWebページを開きます。すると、Paste your input hereと書かれたフォームが表示されますので、CIFを作成したい化合物の座標をコピーします。通常はディフォルトのオプションのままでいいと思いますので、そのまま「Run PRODRG」をクリックして実行します。
すぐ下には「最大2分ぐらいかかるぞ」と書いてありますが、30秒もかからずに結果が出力されます。いろいろな形式の出力ができますが、REFMAC5形式のもの(これがCIFです)を選択してファイルにして保存します。
これで必要となるCIFファイルが取得できました。
2. MAKECIFでCIFファイルを作成する
PRODRGを使わなくてもPDBファイルからCIFを作成することができます。CCP4パッケージのMAKECIFを使います。ここの例ではTRE.pdbというファイルからTRE.cifを作ります。
% makecif --- MAKECIF --- /Vers 5.5.1 ; 12.07.2006/ Do you want to have FILE-DOCUMENT /makecif.doc/ ? /<N>/Y/A : N - means without DOC-file Y - with new contents A - means to keep old contents and add new information with DOC-file program creates batch file: makecif.bat _DOC: (Enter) # input-coord-file FILE_C:TRE.pdb <-- モノマーのファイル名を入力 (省略) #--- type "keyword parameters" and/or --- #--- press key "CR" to run program --- --> (Enter) (省略) I am writing new library: new.lib
ってことでnew.libファイルにRestrain情報を持ったCIFが書き込まれます。その他にもいくつかファイルができるので新しいディレクトリで行う方がいいかもしれません。new.libをTRE.cifとでも名前を変えて保存します。
COOTで使う
COOTを起動したら、Import CIF dictionary...を選択するとCIFファイルの選択ダイアログが出てくるので、先ほど保存したCIFファイルを指定して読み込みます。
これで読み込ませた化合物に対してRegularizeなどが使えるようになります。
残基が飛んで行く・・・ -- 0.4.1
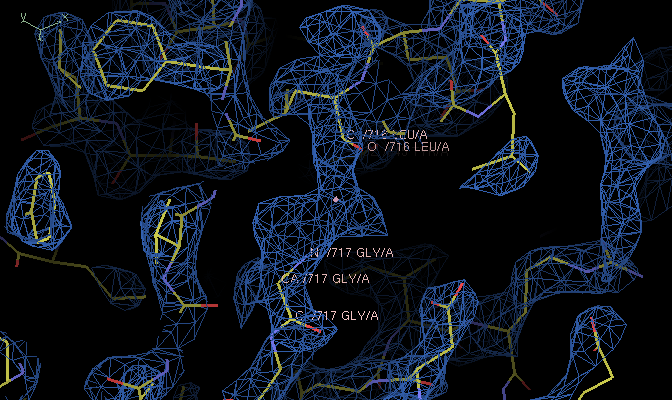
残基の追加や削除を繰り返すようなモデル構築をしているとき、電子密度に原子が合っているように見えるのに残基がばらばらになってしまうことがあります。
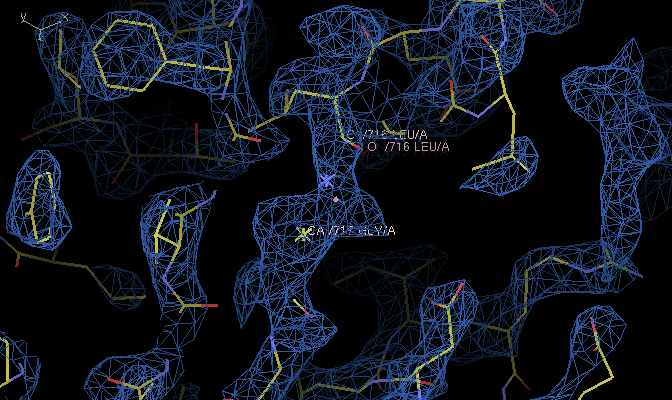
上の図のように配置して、GLY717に対してReal space refinementを実行すると、右図のように残基が崩れます。
ちょっと見にくいですが、よく見ると残基同士が離れているのに残基番号が続いています(716と717)。そこでReal space refinementを実行するとCOOTは続き番号なので残基をつなごうとして崩れるようです。
その場合、Calculate->Renumber residuesで残基番号をずらすと、そのようなバイアスから解き放たれるので問題が解決すると思われます。もちろん、電子密度がイマイチならイマイチですが・・・