PyMOLの使い方
お絵描きプログラムの決定版、PyMOLの使い方を研究してみます。このプログラムは高機能な上に、PyMOLコマンドラインを使うのでとっつきにくいですが、基本を学べばそんなに難しくないと思います。このページではVer.1.1を基にして解説しています。
- チュートリアル -- 電子密度を描くまでのチュートリアル
- 動画の作成 -- 振動や回転といった簡単な動画を作成する手順
- 表面電荷を描く -- APBSを使って表面電荷を描きます
- CAVER -- キャビティ表示のCAVERプラグインのインストール
- スクリプト機能を使う -- PyMOLでスクリプトを実行してみます
- ダイアログボックス -- GUIを作成する基本
- メソッド研究 -- ヘルプのないメソッドを調べた記録(メモ)
- 関連リンク
- Robert Campbell PyMOL Script repository -- PyMOLのスクリプトが置いてあります
基本操作
PyMOLは以下のExternal GUIとViewer + Internal GUIウィンドウで構成されています。
External GUI
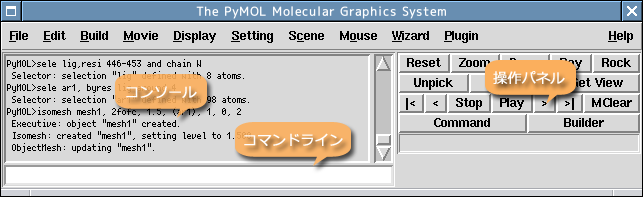
操作メニューなどはここから行います。またコマンドラインやコンソールも備えています。PDBファイルやマップの読み込み、イメージの保存、各種設定などを行う際に使います。
Viewer + Internal GUI
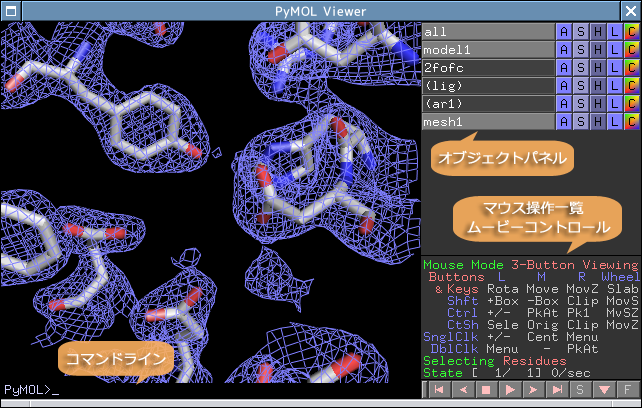
ビュワーとオブジェクトを操作するパネル、フレーム情報などで構成されています。図を描くときにはオブジェクトパネルとコマンドラインを駆使することになります。
オブジェクトパネルに表示されているのは、座標オブジェクト、選択オブジェクト、マップなどがあります。そのうち、選択オブジェクトはselectコマンドで選択された原子の集まりです。描画の制御はオブジェクト名の右側のボタンで行います。
| A | Action | オブジェクトを中心にしたり、削除したりする |
|---|---|---|
| S | Show | オブジェクトの描画方法を指定する |
| H | Hide | 指定した描画を非表示にする |
| L | Label | 描画した残基にラベルを付ける |
| C | Color | 色に関する設定を行う |
記述方法
このドキュメントではPyMOLコマンドラインへの入力を、
PyMOL> select sel1, resi 167
というように表記します。
Selection式 -- selection-expression
原子を選択するときに使用する式です。よく使うと思われる一部を載せておきます。なお、詳しい説明は
PyMOL> help selections
で確認できます。
PyMOLのドキュメントでselection-expressionと示されている部分にはこの式を使います。
セレクタ
選択の基本となる単位です。このセレクタと後述する演算子で原子を選択していきます。対象とするオブジェクトを指定する場合はオブジェクト名を直接指定します。
| 識別子 | 説明 | 例 |
| name <atom names> | 原子名 | |
| resn <residue names> | 残基名 | |
| resi <residue identifiers> | 残基番号 | |
| chain <chain ID> | Chain ID | |
| segi <segment identifiers> | セグメントID(SEGID) | |
| elem <element symbol> | 原子種 | |
| ss <structure> | 二次構造(h,s,l) | ss h+s+l |
| b <>= <b-factor> | 温度因子 | |
| q <>= <occupancy> | 占有率 | |
| ps. <sequence> | シークエンスによる選択 | ps. LYK |
残基名や残基番号は列挙することができます。コンマを含む式には括弧が必要になります。
(resi 91:93,105,270) -- 残基番号91〜93と105、270を選択 (resn TYR,PHE) -- 残基TYRとPHEを選択
演算子
selectionを組み合わせて複雑な選択を行うことができます。
| 論理演算 | |
|---|---|
| selection and selection | 論理積(AND) |
| selection or selection | 論理和(OR) |
| not selection | selection以外 |
| 集合 | |
| selection1 in selection2 | selection1内のselection2 |
| selection1 like selection2 | selection1内でselection2の原子名または残基番号が一致するもの |
| 距離による選択 | |
| selection gap <distance> | selectionのvan dar Waals半径とvan dar Waals半径の距離がdistanceÅある原子 |
| selection around <distance> | selectionの周囲distanceÅの原子 |
| selection expand <distance> | selectionとその周囲distanceÅの原子 |
| selection1 within <distance> of selection2 | selection1に含まれるselection2からdistanceÅの原子 Prot1 within 4 of (resn GLC and Prot2) Prot2のGLCの周囲4Å以内のProt1が選択される。例えばGLCの周りの接触面のSurfaceを描きたい場合に使う。 |
| neighbor selection | selectionに結合している原子 |
| 拡張 | |
| byres selection | selectionが含まれる残基 例) select sel1, byres (resi 10 around 4) -- 残基番号10から距離4Åの残基 |
| byobject selection | selectionが含まれるオブジェクト |
- 複雑な例
- (byres (Prot1 within 4 of (resn GLC and Prot2))) -- Prot2で定義されているGLCの周囲4Å以内のProt1を残基単位で選択
選択マクロ(Atom Selection Macros)
選択マクロを使用することで原子の選択をスラッシュで区切ったシンプルな書式で行うことができます。例えば、
PyMOL> select catalytic, /protA/cat/a/241/ca
とすると、
PyMOL> select catalytic, protA and segi cat and chain a and resi 241 and name ca
と同じ効果があります。書式は以下の通りです。
/オブジェクト名/SEGID/Chain ID/残基番号/原子名
このようになってますが、前および後ろは省略できるようです。つまり、
Chain ID/残基番号/原子名 /オブジェクト名/SEGID/Chain ID/残基番号 Chain ID/残基番号
というように使用することができます。もちろん、残基範囲の指定(12-18など)も使用することができます。
SEGIDはあまり使う人はいないと(勝手に思ってますが)思いますが、CNSなどで使うATOMレコード末尾の部分で最大4文字まで指定できるそうです。まとめて選択するときに便利かもしれません。使わないときは省略可能です。
PyMOL> select catalytic, /protA//a/12-18
というようにスラッシュを続ければ省略できます。
例
PyMOL> zoom resi 241 and chain A -- Chain Aの残基番号241にズームする PyMOL> select sel1, (resi 241-243 and chain A) or (resi 12 and chain B) -- Chain Aの241-243とBの12をsel1という名前で選択 PyMOL> select mol1-A, chain A and mol1 -- オブジェクトmol1のChain Aをmol1-Aという名前で選択
電子密度図を描いてみる
PyMOLでは簡単に電子密度図を描くことができます。必要なものは以下のファイルです。
以上が揃ったらファイルを読み込んでおきます。ここではマップオブジェクトを2fofcとします。
- PDBを読み込んで電子密度を描くまでの手順はチュートリアルで解説しています。
メッシュで電子密度図を描く
isomeshコマンドを使います。
isomesh name, map name, level [, (selection) [, buffer [, state [, carve]]]]
- パラメータ
- name -- メッシュオブジェクト名
- map name -- 使用するマップ名
- level -- 電子密度のσレベル
- selection -- 図を描く基準となるSelection。括弧で囲みます。Selection式でもSelection名でも可。
- buffer -- 描く範囲。たぶん選択エリア + bufferÅ
- state -- 既にあるメッシュオブジェクトを読み込む(1)か、新規作成(0)か。省略時=1
- carve -- 図を描くときの原子からの半径Å
- 例
PyMOL> isomesh mesh1, map, 1.5, (resi 241 and chain A), 1, 0, 2
- 解説
- マップを描く範囲はbuffer, carveで調節できます。ちなみに、buffer=1だけで描いたものとbuffer=1, carve=2で描いたものは以下のようになりました。
これらの値を調節することで余計な電子密度が描かれなくなります。
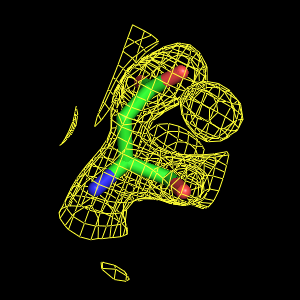
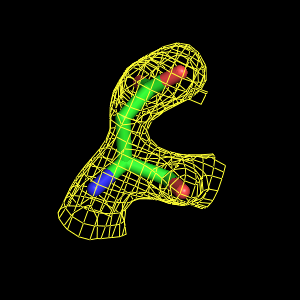
buffer = 1 buffer = 1, carve = 2
動画を作成してみる
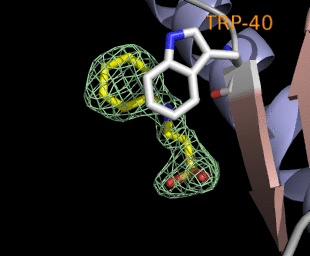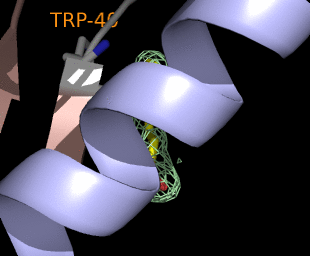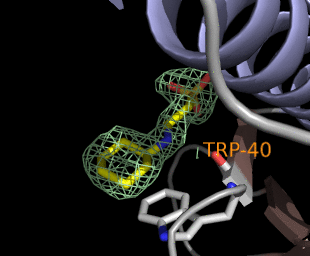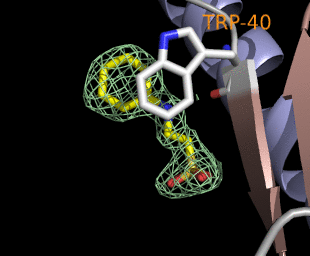
PyMOLは動画も簡単に作れます。右の図はX軸とY軸で回してみたものです。はじめの80フレームでY軸で1周させて続く80フレームでX軸で1周させています。
一連のコマンドは、
mset 1 x160 movie.roll(1,80,1) movie.roll(81,160,1,"x")
で作成できます。全部で160フレームなのでレンダリングすると結構時間がかかります(まあ、画像のサイズが小さいので10分程度ですが)。
詳しくは、
を参考にしてみてください。
斜め回転もさせてみたいのですが・・・Pythonスクリプトが必要かな?
