- PyMOLクックブック
- 水素結合などの点線を描きたい
- CA(Cα)のみのPDBファイルを表示したい
- 微妙な結合距離のためか、共有結合が切れている
- 点線のパラメータを調整したい
- 一部の残基のみスティックの太さを変えたい
- セルを描きたい(作成中)
- オブジェクトを半透明にしたい
- Sphereの半径を指定したい
- 立体視用イメージを作成したい
- スナップショットを保存したい
- 表示領域のサイズを設定/取得したい
- 任意のラベルを置きたい
- ラベルのサイズ、相対位置を調整したい
- 正確にカメラ方位を変更したい
- オブジェクトを回転させたい
- 同一PDBファイル内の分子を別オブジェクトにしたい
- 分子を重ね合わせたい
- 特定の原子を基準に分子を重ね合わせたい
- 分子を置換したい
- 現在の方位を取得したい
- 二次構造を変更したい
- 変な結合ができる
PyMOLクックブック
PyMOLでお絵描きするための小技集。美麗なイメージを目指せ!
コマンドの文法表記は以下のような表記規則に従います(たぶん)。
| 表記例 | 意味 |
| 太字 | PyMOLコマンド |
| 透明度(0-1) | 値を指定する。カッコ内は値の範囲など |
| {H|S|L} | どれかを選択(これならH,S,Lのどれか)。 その他、dash_{gap|length}ならdash_gapとdash_lengthってこと |
| [, mode] | 省略可能なパラメータ |
2011-06-10 -- いつの間にか数が増えてきたので徐々にジャンルごとに分けていこうと思います
- 表現方法 -- 2011-06-10 (金) 14:16:03
水素結合などの点線を描きたい
- 問題
- 水素結合などを示す点線を描きたい
- 解法
- distanceを使います。
名前指定でオブジェクト名を付けることができます。省略した場合はdist01,dist02...になります。また、カットオフ値を指定することで表示する最長の距離を指定することができます。モードはなんだかわかりません(ディフォルト0)。distance 対象1, 対象2
distance 名前 = 対象1, 対象2 [, カットオフ [, モード]] - マウスで選択することで作成します。
Ctrl+真ん中ボタンで線を引きたい2つの原子を選択した後、コマンドラインでdistと入力します。
- 参考
CA(Cα)のみのPDBファイルを表示したい
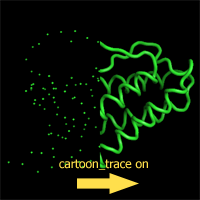
2010-12-16 : 追加
- 問題
- FATCATで取得したPDBファイルはCαのみなので原子しか表示されない
- 解法
- cartoon_traceを真にセットします。
通常メニューからではCαだけではトレースされないので以下のコマンドでトレースします。PyMOL> set cartoon_trace, 1 PyMOL> show cartoon
これでFATCATの.morph.pdbファイルも表示できます。
微妙な結合距離のためか、共有結合が切れている
2010-04-13 : 追加
- 問題
- 共有結合を作りたい
- 解法
- bondを使います。
distanceと同様に対象となる2つの原子を共有結合で結合します。対象はCtrl+真ん中で選択することも可能です。bond 対象1, 対象2
なお、結合を削除する場合はunbondを使います。
点線のパラメータを調整したい
2010-01-27 : 色と対象について更新
- 問題
- 点線を描いてみたが点の間隔が気に入らない
- 解法
- set dash_gap, set dash_length, set dash_widthで調整します。
例えばこんな感じ。set dash_{gap|length|width}, 値[, 対象]
PyMOL> set dash_gap,0.2 PyMOL> set dash_length,0.2 PyMOL> set dash_width,2 PyMOL> set dash_color,tv_blue
rayコマンドでレンダリングするとギャップが狭くなるので確認して下さい。なお、複数選択の場合は以下の感じで。PyMOL> set dash_color,tv_blue,dist01:dist02 PyMOL> set dash_color,tv_blue,dist* <-- ワイルドカードも使えるようです
- 参考
一部の残基のみスティックの太さを変えたい
2010-09-08 : 作成
- 問題
- 一部の残基の太さのみを変更したいが、set stick_radiusコマンドでは全体しか変更できない
- 解法
- createおよびset stick_radiusを組み合わせます。
以下のような感じで行うとできます。PyMOL> create forSticks1, resi 1-2 PyMOL> show sticks, forSticks1 PyMOL> set stick_radius, 0.15, forSticks1
- 参考
セルを描きたい(作成中)
- 問題
- 単位格子を表す枠を描き、色指定および立体的に描画したい
- 解法
- 単位格子を表すセルを描いてみます。任意の場所に出現させるのは現在調査中・・・
この例ではセルを描いて色を変更したりレイトレーシングの時に立体的に描画するようにしています。 まずPDBファイルを開いておきます。開いたPDBのオブジェクト名はmol1とします。以降はスクリプトで。PyMOL> show cell,mol1 セルを表示 PyMOL> color red,mol1 色を赤にしてみる PyMOL> util.cbag mol1 分子だけ元の色に戻す PyMOL> set cgo_line_width=3 セルの線の太さを変更 PyMOL> set cgo_line_radius=0.4 3D描画の際のセルの線の半径を変更 PyMOL> ray レイトレース
これはCompiled Graphics Objects(CGOs)という機能を使っているようです。詳しいことはドキュメントにも書いてませんが・・・
- 参考
オブジェクトを半透明にしたい
2011-01-28 : 更新
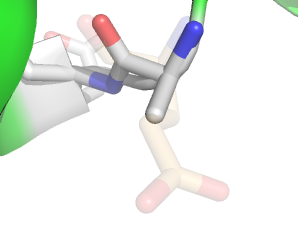
- 問題
- 残基の側鎖などを重ねて描くときに、一方を半透明にしたい。
- 解法
- stick_transparencyまたはcartoon_transparency、sphere_transparencyを使います。なお、表面(surface)を半透明にする場合はtransparencyに値をセットします。
これらのコマンドで、対象となるオブジェクトのtransparencyの値をセットします。set stick_transparency, 透明度(0-1)[, 対象]
例えば対象となるオブジェクトがmol1なら、PyMOL> set stick_transparency,0.5,mol1
で半透明になります。stick_ならstickオブジェクト、cartoon_ならリボン図が対象になります。透明度は1が完全に透明、0が不透明です。
右の例ではstick_transparencyを0.8にセットしてレイトレースしたものです。PyMOL 1.1で試したところstick_transparencyでは対象にセレクトオブジェクト名(selectで作ったオブジェクト)を指定しても効果が適用されませんでした。分子名で指定する必要があるみたいです(たぶん)。sphere_transparencyではセレクトオブジェクトでも半透明になりました。 - 参考
Sphereの半径を指定したい
- 問題
- sphereで描いた原子の大きさを調整したい。
- 解法
- 描画比率を変更する
van der Waals半径と実際の描画との比率を変更することで原子の大きさを変更します。
PyMOL内部でvan der Waals半径に対する比率が設定できるのでその値を変更します。この方法だと異なる原子のvan der Waals半径の比を保持したままサイズを変更できます。特定の原子の大きさだけを変更したい場合はalterコマンドを使用します。set sphere_scale, スケール[, 対象]
PyMOL> set sphere_scale, 0.2
- van der Waals半径の値を変更する
alterコマンドを使います。
van der Waals半径を指定するという形で調整可能なようです。alter 対象, vdw=VDW半径
実際に使用する時はalterコマンドの後、rebuildして更新します。PyMOL> alter wat, vdw=0.5 PyMOL> rebuild
立体視用イメージを作成したい
- 問題
- 立体視用のイメージを作成したいが、マウスで角度を変えたくない。
- 解法
- rayコマンドにオプションを付けて使います。
レイトレーシングを行うrayコマンドにangleオプションを付けます。角度は大体2-3°ぐらいがよさそうです。3°で深度が強調されるぐらいで、2°ならほどよい気がします。ray angle=角度
PyMOL> ray angle=2 <- たぶん交差法左用 PyMOL> ray angle=-2 <- たぶん交差法右用
なお、右と左は交差法と平行法で逆になりますので注意して下さい。
スナップショットを保存したい
2011-03-08 : 追加
- 問題
- イメージを出力したいがrayコマンド後、メニューから選ぶのが面倒くさい
- 解法
- rayコマンドおよびpngコマンドを使用します。
メニューを使用すれば、rayコマンドを使用した後、Save image asで保存可能(試用版はできなかったかも)ですが、pngコマンドを組み合わせることで簡単に保存できます。PyMOL> ray angle=2; png snapshot_+2.png PyMOL> ray angle=-2; png snapshot_-2.png
という感じです。
なお、pngコマンドではイメージのサイズも指定できますが、rayの後でイメージ指定をするとレンダリングが解除されてしまうのであらかじめ表示領域のサイズを目的のサイズにしておきます(pngのrayオプションではangleが指定できないため)。 - 参照
表示領域のサイズを設定/取得したい
2011-03-08 : 追加
- 問題
- 表示領域のサイズを数値で指定または取得したい
- 解法
- 設定:viewportコマンドを使用します。
画像のサイズを一定にしたい場合、ウィンドウサイズをいちいち合わせていると面倒ですが、viewportコマンドを使えばピクセル単位でサイズを合わせることが可能です。なお、ウィンドウ操作用のwindowコマンドもありますが、これでサイズを変更するとコントロールパネルを含んだサイズになるため表示領域のサイズ指定はできないようです。viewport width=幅(px), height=高さ(px)
PyMOL> viewport 800, 600 <-- 800x600のサイズになる
- 取得:cmd APIから取得します
取得は若干面倒でPyMOL APIを使うことになるようです。以前のバージョンではコマンドが存在していたようですが削除されたとのこと。PyMOL> print cmd.get_session()["main"] [837, 1074] <-- 幅、高さ(px)で取得できる
- 参照
- [PyMOL] Access viewport size -- http://www.mail-archive.com/pymol-users@lists.sourceforge.net/msg08419.html
任意のラベルを置きたい
2011-01-28 : 追加
- 問題
- 任意の場所にラベルを置きたい。
- 解法
- 右クリックで現れるMain Pop-upメニューから[new]->[pseudoatom]->[label]で追加できます。
オブジェクトに表示されるラベルでは自由な文字列が表示できませんが、Main Pop-upメニューからのコマンドで任意の文字列をラベルとして追加することができます。なお、ラベルの位置はEditingモードでCtrl+左ドラッグで移動することができます。Pseudoatomとなっているだけあって通常の原子と同じように移動させることが可能です。
ラベルのサイズ、相対位置を調整したい
- 問題
- 残基名や距離などのラベルの位置や大きさを変更したい。
- 解法
- External GUIからセットする
PyMOLではオブジェクトにラベルを表示させることができますが、表示させたラベルのパラメータはExternal GUIの[Setting]->[Edit All...]から調整します。このメニューを選ぶとパラメータが並んだダイアログが開きますので必要なパラメータを変更します。
このダイアログ中では全てのパラメータが表示されて探すのが大変なので、下にあるFilterにキーワードを入力して絞りこみます。例えば今回はラベルの関連なのでlabelと入力しして絞り込んでみると、関連すると思われるパラメータが表示されますので、その中から必要と思われるパラメータを探して値を変更します。値を変更した後にEnterキーを押さないと反映されないので注意!
主要なパラメータを下にまとめておきます。 - コンソールでセットする
コンソールで直接セットすることもできます。PyMOL> set label_size, 25
適切な値は表示を見ながら合わせて下さい。
表示を調整するための主要パラメータ
| パラメータ名 | 内容 | 設定例 |
| label_color | ラベルの色 | white, orangeなど |
|---|---|---|
| label_font_id | フォントの種類 | 数値(環境依存?) |
| label_outline_color | 縁取りの色 | white, orangeなど |
| label_position | オブジェクトからの相対位置 | [x,y,z] |
| label_size | フォントサイズ | 数値 |
他にもいろいろパラメータはありますが、だいたい名前を見れば想像できると思うのでいろいろ試してみて下さい。
ラベル以外のパラメータの中には[Setting]メニューから簡単に設定できるパラメータもあるので確認してみて下さい。
正確にカメラ方位を変更したい
2010-01-14追加
- 問題
- 例えばY軸に対して90度方位を変えた図を描きたいが、マウス操作では正確ではない。
- 解法
- turnメソッドを使います。
turnメソッドにより視点を回転させることができます。例えば90度横からの図を描きたい、などに有効です。以前、rotateでできると書いていましたが、それだとオブジェクトが回転されてしまうので目的とは異なってました・・・turn 軸, 角度
PyMOL> turn y, 90
Y軸(画面の中心)を基準に90度回転させる。
オブジェクトを回転させたい
2010-01-14修正
- 問題
- 特定のオブジェクトのみを回転させたい
- 解法
- rotateメソッドを使います。
rotateメソッドを使うと回転角度を数値で指定することができるので正確に回転させた図を描くことができます。メソッド中のカメラ、オブジェクト、座標(origin)については詳しくは調べてません・・・回転中心などを指定できるのだと思いますが。rotate 軸, 角度[, 対象[, カメラ[, オブジェクト[, 座標]]]]
PyMOL> rotate y, 90
Y軸(画面の中心)を基準に90度回転させる。
同一PDBファイル内の分子を別オブジェクトにしたい
2011-01-07 : 追加
- 問題
- 例えばタンパク分子の表面モデルを描き、リガンドをスティックで描きたいとき同一PDBファイルだと表面がうまく描けない
- 解法
- createメソッドを使います。
一つのPDBファイルにタンパク分子とリガンド分子がChain AとChain Bで記述されていた場合、PyMOLのsurfaceコマンドで描くとChain AとChain Bの境界面が描画されません。それはたとえセレクションを使って別々に描いても同じです。
この問題を解決するには、タンパク分子とリガンド分子のオブジェクトを分割すればいいのですが、PDBファイルをわざわざ別々にするのも手間がかかります。そこでcreateで読み込んだPDBから別々のオブジェクトを作成します。
ここではpdb_nameが読み込んだPDBファイル、Chain Aがタンパク分子、Chain Bがリガンド分子だとします。PyMOL> create Protein, chain A and pdb_name PyMOL> create Ligand, chain B and pdb_name
これでProteinとLigandという名前を持つ新しいオブジェクトが作成されました。またはpdb_nameオブジェクトからLigandオブジェクトを作成し、removeでpdb_nameからLigandオブジェクトを削除してもOK。PyMOL> create Ligand, chain B and pdb_name PyMOL> remove chain B and pdb_name
こうすると、タンパク分子だけが残ったpdb_nameオブジェクトとLigandオブジェクトが作成されます。
メソッドの文法は以下の通り。create 名前, 対象
remove 対象
分子を重ね合わせたい
2011-01-20 : 追加
- 問題
- 分子同士を二次構造で重ね合わせたい
- 解法
- alignメソッドを使います。
単純な重ね合わせ図を描く場合はalignで合わせると楽でいいです。PyMOL> align source, target
で重ねられます。
特定の原子を基準に分子を重ね合わせたい
2010-09-09 : 追加
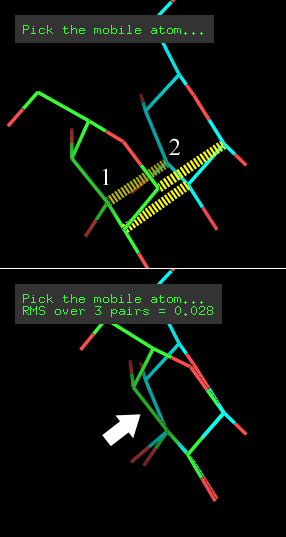
- 問題
- 特定の分子を原子を指定して重ね合わせたい
- 解法
- [Wizard]->[Pair Fitting]を使います。
そのお絵描き機能にばかり目がいってしまいがちですが、案外編集機能もあるPyMOL。Pair Fittingを使うことでフィッティングする原子を指定して特定の分子(例えばリガンド同士など)を重ね合わせることができます。COOTなどでも手作業で合わせることができますが、論文に書くなら、Pair Fittingを使うことで、重ね合わせた基準をしっかり示せる上、RMSDまで求めてくれるので便利だと思います。実は最近知りました。もっと早く知っていれば・・・
使い方は簡単。1.動かしたい分子中の重ね合わせたい原子をクリック、2.重ね合わせ先の原子をクリック、を繰り返すだけです。いくつか原子を指定した後、Fit 3(ペアの数) Pairsをクリックすると重ね合わせてRMSDを計算してくれます。私が試したとき、1 Pairだけだと分子が消えてしまう事象(バグ?)が発生しましたが、2 Pair以上だと問題は起こりませんでした。
分子を置換したい
2010-09-30 : 追加
- 問題
- 既に残基などを選択している分子があるが、少しいじったので更新したい
- 解法
- updateメソッドを使います。
例えばある程度絵を描いたところで(書きたい残基などを選択したりして)、「GlnやAsnの酸素と窒素が逆のほうがよかった!」という経験はよくあると思います(わたしだけ?)。そんなときに新たな分子を読み込み、選択状態などを保持したままその分子と置換できれば・・・というときに使うコマンドです。
使い方は至って簡単。PyMOL> update old_molecule, new_molecule
です。old_moleculeは更新したい分子、new_moleculeは更新後の分子です。このコマンドを実行することで状態を保ったまま置換することができます。ただ、distなどの線は原子が動いても追随してくれないようなのでその辺りは再度作成する必要がありそうです。
現在の方位を取得したい
2009-12-22追加
- 問題
- 向きを変えて構造を確認したいが、現在の描画の方位を変更したくない。
- 解法
- External GUIウィンドウのGet Viewボタンをクリックするか、get_viewメソッドを使います。
現在の方位を一度記憶してからモデルを確認するという方法が使えます。方位を取得すると現在の方位パラメータが得られるので、その値を使ってset_viewコマンドで戻すことができます。PyMOL> get_view ### cut below here and paste into script ### set_view (\ 0.317731529, 0.080384687, 0.944767177,\ 0.366989166, -0.929166734, -0.044363577,\ 0.874280035, 0.360815048, -0.324725896,\ 0.000000000, 0.000000000, -446.314361572,\ 34.259254456, 0.657300949, 33.577735901,\ 351.877655029, 540.751098633, -20.000000000 ) ### cut above here and paste into script ###
このような出力が得られるので、この部分をコピーしてset_view()を実行すれば元の方位に戻ります。
二次構造を変更したい
- 問題
- DSSPで計算した二次構造とPyMOLで表現される二次構造が異なっている。
- 解法
- alterメソッドを使います。
DSSPなどの二次構造計算プログラムで計算した結果とPyMOLで自動的に割り当てられる二次構造が異なることがあります。これは構造を詳しく議論したい場合に問題となることがありますので図を合わせておきたいところです。そんなときはalterメソッドを使うことで修正することが可能です。
alterはいろいろな事を変更できるようですが、ここでは二次構造を変更します。Hはへリックス、Sはストランド、Lはループを表します。alterだけでは二次構造が書き換わらないので実行した後rebuildを使用します。alter 対象,ss='{H|S|L}'
PyMOL> alter (resi 23-25 and chain A),ss='S' PyMOL> rebuild
これでChain Aの残基番号23-25がストランド表現に変更されます。
変な結合ができる
2009-12-22追加
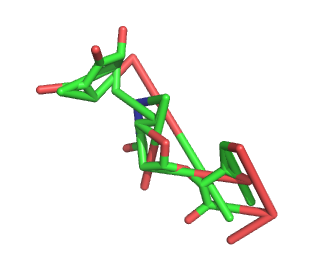
- 問題
- 右の図のように変な結合ができてしまい何だかわからないコトになっている。
- 解法
- PDBファイルのCONECTレコードを削除します。
PDBの内容にもよると思いますが、CONECTレコードが悪さをしていることがあるようです。あまり細かくは検証していませんが、CONECTレコードは原子の連番と結びつけられているようなのでその番号が変わってしまっている、などが考えられます。うまく使えばつながらない結合をつなぐこともできるかもしれません。
