チュートリアル
実際に使い方に慣れるためにPyMOLを使って残基に2Fo-Fcマップを表示させてみます。目指す図は、バックボーンはリボン図、表示させたい残基をスティックで表示してそれに電子密度を重ねるという単純なものですが、PyMOLで絵を描く基本動作なので理解すれば応用することが可能だと思います。
ファイルの読み込み
まずはファイルを読み込みます。必要なファイルは、1) PDBファイル、2) 2Fo-Fc電子密度図です。電子密度図はXPLOR(CNS)フォーマットまたは、CCP4、BRIX(O)フォーマットが使用可能なようです。
参考までに今回使用した、PDBファイルと作成したマップファイルはこれです。PDBから取得して作成しました。
 2dsy.pdb -- PDBファイル
2dsy.pdb -- PDBファイル 2fofc.ccp4.bz2 -- CCP4形式マップファイル(3MBほど;bzip2圧縮してます)
2fofc.ccp4.bz2 -- CCP4形式マップファイル(3MBほど;bzip2圧縮してます)
まずPDBファイルを読み込みます。External GUIメニューの[File]->[Open]で読み込みます。ファイル選択ダイアログから適当なPDBファイルを選択してください。続いて同様に、2FoFcマップを読み込みます。CCP4形式のマップファイルなら拡張子.ccp4、XPLORなら.xplorにしておく必要があるので注意してください。これらのファイルを読み込んだ後、PDBファイルと2fofcファイルがオブジェクトパネルに表示されているはずです。
この例ではPDBから取得した2DSYを用いて話を進めます。
全体のリボン図
分子の色分け
このタンパクではNHEという分子が配位しているのが観測されていますので、そのあたりの電子密度を描くことを目標にしてみますが、まず、基本的な図を描くことから始めます。このタンパクは非対称単位中に4分子入っていますので、これらを色分けして表示させます。
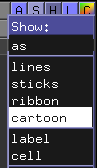
オブジェクト2dsyの[H]をクリックするとHide:ウィンドウが開くので、[everything]をクリックするとタンパク全体が消えます。これは実際に消されたのではなく、表示されていないだけですのでご安心を。続いて、[S]をクリックしてShow:ダイアログから[cartoon]をクリックしてください。これで全ての分子がリボン図で表示されたと思います。
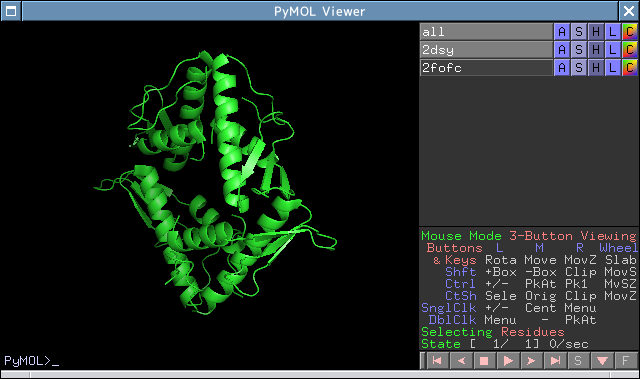
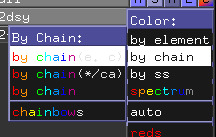
リボン図になったら分子ごとに色を変えてみましょう。2dsyオブジェクトの[C]をクリックするとColor:ダイアログが表示されますので、その中の[by chain]->[by chain(e. c)]をクリックします。これで色分けして表示されます。適当な角度にしてイメージとして保存しましょう。
イメージの保存とレイトレーシング
イメージの保存はExternal GUIの[File]->[Save Image As]->[PNG]で行います。これで表示された通りのイメージがPNG形式で保存されました。ただ、この図は編集用の描画なので、よく見るとギザギザが目立ったり影がなかったりしてちょっと荒いです。論文などに使うような高品質なイメージにするにはレイトレーシングを行います。レイトレーシングはPyMOL内部のレンダラまたはPOV-Rayを用いて行えますが、ここではPyMOL内部レンダラを用います。
まず、画質を選択します。External GUIの[Display]->[Quality]のオプションで速度優先か画質優先かを選択することができます。最高画質にしたいなら[Maximum Quarity]、速度優先なら[Maximum Performance]を選択します。このように描画などのオプションはExternal GUIで行えることが多いので(コマンドラインを使うこともありますが)、いろいろいじってみるといいかもしれません。
オプションを選択した後、レイトレーシングをします。External GUIの下にある入力ボックスまたは、Viewerの下のPyMOL>の行、PyMOLコマンドラインを使います。External GUI、Viewerのどちらのコマンドラインでもいいので、
ray
と入力してEnterを押すとレイトレーシングが始まります。
今後、このようにコマンドラインを使う際は、
PyMOL> ray
と表記しますので覚えておいてください。この表記中のPyMOL>はコマンドプロンプトを表すので入力しないようにしてください。また、PyMOLでは大文字小文字は区別するようなので正確に入力してください。
しばらくするとレイトレーシングされた高品質な画像が表示されると思います。ただし、ここで分子を動かしてしまうと表示が戻ってしまうので描画が分子を操作せず、[File]->[Save Image As]->[PNG]で保存しておきます。
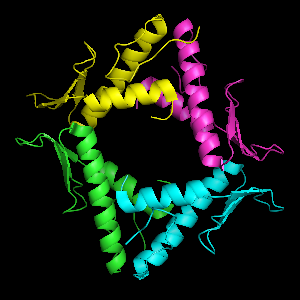 | 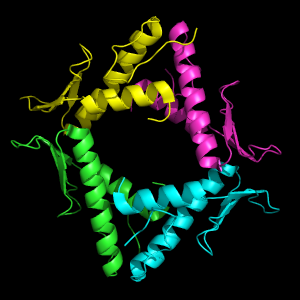 |
| レイトレーシングなし | レイトレーシングあり |
|---|
単量体を描いてみる
単量体の描画
全体図が描けたので単量体を描いてみることにします。まずChain Cを選択して、その分子を中心に持ってきます。コマンドラインから、
PyMOL> select molC, chain C
でChain Cを選択します。原子を選択する際の文法はPyMOLを使ってみようのSelection式を参照してください。
選択されたらオブジェクトパネルに(molC)という項目が追加されたと思いますが、この(molC)の[A]を開いて[center]をクリックすると中心がChain Cに移ります。後は先ほどと同様に全体を消して、molCを表示させます。
まず、2dsyの[H]->[everything]、続いて(molC)の[S]->[cartoon]で表示されます。選択されている状態表示が邪魔な時は(molC)と表示されている部分をクリックすれば表示が消えます。
これで、単量体が表示されましたが、単色では芸がないので少しアレンジします。(molC)の[C]で色を変更できるので適当に変化させてみましょう。
化合物の表示
実はこのChain Cには低分子が配位しているので表示させます。
PyMOL> select nhe, resi 1001
で選択すると(nhe)がオブジェクトパネルに追加されますので、[S]->[sticks]で表示させると化合物が表示されます。色は、[C]->[by element]で原子種ごとに色を設定することができます。
二次構造での色分け
化合物も表示されましたがまだ平凡なので、ここでヘリックスとストランドの色を変えてみます。[C]->[by ss]でも色分けすることが可能ですが、他の方法を紹介します。二次構造を指定することで選択して色を付けます。
PyMOL> select hel, ss h and chain C PyMOL> select str, ss s and chain C
これでヘリックスのみの(hel)、ストランドのみの(str)オブジェクトができたはずです。後は同様に[C]で色を変更します。お気に入りの図ができたらレイトレーシングして保存してください。ちなみに、背景の色はExternal GUIの[Display]->[Background]で変えることができます。
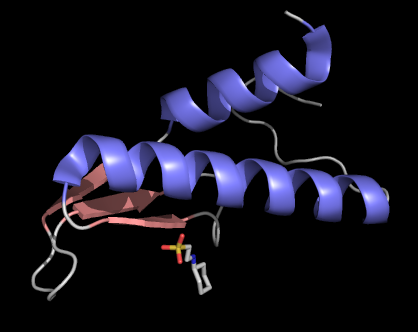
電子密度の表示
基本的な分子の描画ができたので電子密度を重ねてみます。ここまできたら電子密度図を描くのは簡単です ![[smile]](image/face/smile.png) 。
。
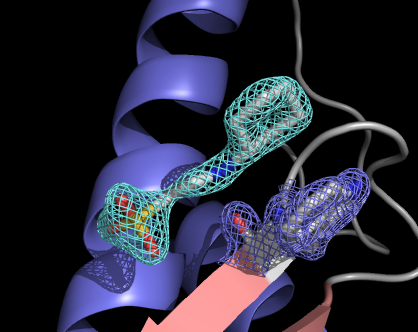
この分子はNHEとTRP40がスタッキングしているのでそのあたりを描いてみます。
PyMOL> isomesh mesh1, 2fofc, 2, (nhe), 1, 0, 2 <-- NHEの電子密度を描く PyMOL> select w40, resi 40 and chain C <-- TRP40を選択 PyMOL> isomesh mesh2, 2fofc, 2, (w40), 1, 0, 2 <-- TRP40の電子密度を描く
このままではTRP40の側鎖が描かれていないので、(w40)の[S]->[sticks]で表示させます。最後に(nhe)を[A]->[center]で中心に持ってくれば完成!
なお、電子密度mesh1とmesh2も[C]で色を変更できるので試してみてください。