もっとCOOT!
基本のキから急に飛びますが、COOTのもうちょっと実践的な使い方をメモしていきます。
古いバージョンだと機能が不足している場合があるかもしれないので試したバージョン番号も記述しておきます(示したバージョンより古い場合でも使えるものもあると思います)。最近のCOOT(バージョン0.5.2以降)は機能がガシガシ追加されていってますのでメニュー項目が整理しきれてない気がします・・・バージョンによって項目の場所が違うかもしれないので注意!
Dynamic Distance
- 0.6pre1 build1716
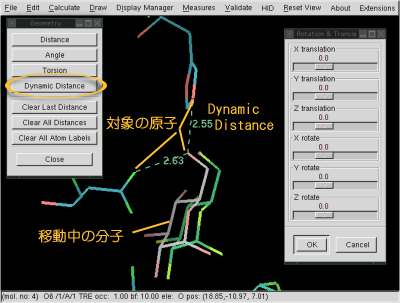
例えばリガンド結合させたモデルを作るとき、明確な電子密度があればよいのですが、電子密度もはっきりしない状態でモデルを組みたい時ってありませんか? 特に論文で結合様式モデルの図を作成する際にリガンドを置くような時、そんな状況で役に立つのがDynamic Distanceです。
通常、Environment Distancesなどを使ってもRotate/Translate Zoneでモデル修正をするときには距離の値は変わりませんが、この機能を使うと、修正しながら距離を確認することができます。使い方に癖がありますが、以下のように使います。
- [Measures] -> [Distances & Angles]ダイアログを開く
- [Calculate] -> [Model/Fit/Refine]ダイアログの[Rotate/Translate Zone]を選択
- 通常どおり動かしたい残基、リガンドを選択 -> 分子が、移動できる白い表示になる
- [Distance & Angles]ダイアログの[Dynamic Distance]をクリックで選択状態に入ります
- 対象となる2点の原子をクリック。この時、一方は移動中の分子内の原子です
- 分子の移動にあわせて距離が表示される(複数の距離も表示可能です)
ポイントは、「Rotate/Translate Zoneでの移動中の分子中の原子を選択する」ということです。移動中状態でない場合は選択できません。試していませんが[Real Space Refine Zone]中などでも使えるかもしれません。
Other Modelling Tools
ver.0.6 2010-01-06
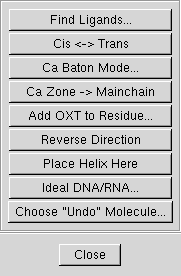
[Calculate] -> [Other Modelling Tools...]でOther Modelling Toolsダイアログが開きます。
0.6よりFind Waters、Add OXTなどがこちらに移動しました。その他、以前のバージョンでは分散していた機能が、まとめて使えるなったものです。今後のバージョンではこのあたりが充実してくる予感。新しく追加された機能は、Find Ligands、Cis <-> Trans、Ca Baton Mode、Ca Zone -> Mainchain、Add OXT to Residue、Reverse Direction、Place Helix Here、Ideal DNA/RNA、Choose "Undo" Moleculeのなどがありましたので、いくつか試してみました。
Find Ligands
リガンドと思われる電子密度を探してくれます。この機能を使うためにはあらかじめリガンド分子を読み込んでおく必要があります。設定できる項目は、
- 対象となるマップ
- マスクとして使用する分子(通常はタンパク分子ですね)
- 既に置いてある水分子をマスクとするか無視するか
- 検索するリガンド分子(複数可のようです)とFlexible検索を行うか
- マップのシグマレベル
です。既に置いていたリガンド分子で試してみたところ、2つ見付けて、ひとつはまあまあの位置(もともとが怪しいのですが)、ひとつはヘンなところでした。また、分子構造をFlexibleとして検索できますが、これには分子のCIFライブラリが必要になりますので特殊な場合はMAKECIFで作成してください。Flexible検索は単純検索に比べて少し時間がかかるようです。
Cis <-> Trans
ペプチド結合のCisとTransを変換することができます。Model/Fit/RefineのFlip Peptideよりも大きく変化します。
Place Helix Here
対象となる電子密度にヘリックスを置きます。どの程度役に立つのかは不明 ![[huh]](image/face/huh.png) 。
。
なおAdd Strand Hereコマンドが[Extensions] -> [Modelling]にあるところが開発途上な感じがします ![[smile]](image/face/smile.png) 。
。
バックアップファイルからの復活
- 0.6pre1 build1716
COOTを使用していると突然プログラムがクラッシュすることがあります ![[sad]](image/face/sad.png) 。この際のダメージを最小限に食い止めるためには時々ファイルを保存することぐらいしかないと思いますが、もうひとつ被害を食い止める方法があります。それがバックアップファイルです。
。この際のダメージを最小限に食い止めるためには時々ファイルを保存することぐらいしかないと思いますが、もうひとつ被害を食い止める方法があります。それがバックアップファイルです。
同じディレクトリでずっと作業をしているとCOOT終了時に「古いバックアップファイルがたまったが削除してよいか?」というようなことを聞かれることがあります。これがバックアップファイル群です。古いファイルは置いておいてもそんなに意味はないと思いますが、最新のものはクラッシュしたときの救世主になります。
モデリングで分子を操作したときごとに現在のディレクトリのcoot-backup以下に保存されます。ファイル名は、
_disk_data_dobuo_mutant_CCP4i_090629-4_refmac1.pdb_Mon_Jun_29_14:51:53_2009_modification_0.pdb.gz
というような名前になるはずです。クラッシュしたときの最後の修正はこれをgunzipで展開してから使えば元に戻すことができるはずです。
・・・とはいってもこまめな保存は習慣づけておいた方がいいのかな・・・?
Check/Delete Watersで効率的に
ver.0.6 2010-01-06
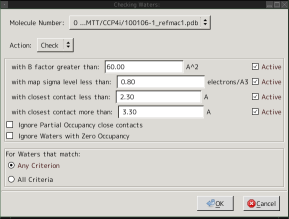
構造の精密化最大の宿敵(?)の水分子のピックアップですが、拾いすぎに気を遣ってませんか?
そんな悩みを吹き飛ばすのが[Validate] -> [Check/Delete Waters]コマンドです。このコマンドは拾った水分子に対して条件を指定することで余分な水分子をチェックまたは削除してしまうという強力なツールです。検索条件は温度因子、マップのシグマレベル、近接原子との最短距離、最長距離を指定でき、必要に応じてパーシャルまたは0のoccupancyを持つものを無視することもできます。もちろんAND/ORにも対応!
参考までに私の使い方の例を載せておきます。
- [Calculate] -> [Other Modelling Tools]の[Find Waters]で水を拾う。データの状況にもよりますが私は大体、Fo-Fcマップを使い、シグマレベルは3、最短距離は2.3Åで最長距離は3.3Åにしています。このあたりはお好みで。
- 続いて通常通りチェックして追加するなり減らすなりします。
- REFMACなどを流す。ここまでを満足できるぐらいまで繰り返します。このあたりは通常通り。
- 水分子が増えてきたらCheck/Delete Watersの出番です。とりあえず、温度因子が60以上(データによって適当に)のものと、2Fo-Fcのシグマレベルが0.8以下のものを削除。いきなり削除するのが怖い場合はCheckしてから。
- それだけでだいぶ余計なものが削除されるはずなので、距離が2.3Å-3.3Åから外れるような水分子をCheckして、不要なら削除していきます。
慣れてしまえば、一定の基準で水分子を拾うことができるのでそこそこクォリティを上げて行けるのでは? お試し下さい。
修正した残基の温度因子をリセットする
ver.0.6.2pre1:2011-05-30
例えば側鎖の向きを変えた場合に温度因子をリセットするには、Residue Infoから変更することが可能ですが、それを自動的に行ってしまう設定があります。スクリプトウィンドウからの実行になりますが、
COOT-Scheme> (set-reset-b-factor-moved-atoms 20)
というようにすれば動かした残基の温度因子の値をリセットすることができます。なお、この設定を行って以降の移動に対して有効です。無効にするには0を指定します(ディフォルト)。
