構造を解析する
得られた構造の解析を行います。
アミノ酸配列のアライメントをしたい
構造情報を含めない
ClustalWを使います。
分子置換で構造を決定した場合、MOLREPやPHASERの処理内で配列を置換することもできますが、手で行う場合に行います。分子置換プログラムでミューテーションしてもいいのですが、結局ひとつずつ見るので私はひとつずつ修正しながらミューテーションしています。
% clustalw2 -infile=sequences.seq -outfile=alignment.aln
立体構造も含めてアライメントする
奈良先端大のMATRAS Webサービスを使います。
立体構造を含めたアライメントを行うことができるので活性残基などを議論するときに有効です。
分子の接触面積を求めたい
CCP4パッケージのAREAIMOLを使います。
ダイマーなどで分子AとBの接触面積を求めたい場合があります。この場合はCCP4のAREAIMOLプログラムを使ってみましょう。
AREAIMOLはタンパク分子の表面上に1.4Åの球を転がして表面積を求めることができるようです。
モノマーだけのアクセシブルエリアを求める
まず、モノマー分子molA.pdbの表面積を求めます。
#!/bin/sh areaimol XYZIN molA.pdb \ XYZOUT sdiff1.brk <<eof-area DIFFMODE OFF MODE NOHOH SMODE OFF OUTPUT END eof-area
こんな感じのスクリプトファイルを作成します。実行するとずらずらっと結果が出ますが、表面積(と思われる部分)は以下のようになっています。
ANALYSIS OF ACCESSIBLE AREAS BY RESIDUE --------------------------------------- GLY A 3 52.00 MSE A 4 178.00 GLY A 5 21.00 ..(略) LEU A 81 74.00 PRO A 82 192.00 <B><FONT COLOR="#FF0000"><!--SUMMARY_BEGIN--> Total area of chain A: 5410.0 -------------------------------------------
この結果によると分子Aの表面積は5,410Ųと計算されます。
ダイマーの表面積よりAとBの接触面積を求める
モノマーの表面積が分かったので、ダイマーの表面積を求めます。先ほどの例のmolA.pdbの部分を分子AとBを合わせたPDBに変えるだけです。その結果は以下のようになりました。
ANALYSIS OF ACCESSIBLE AREAS BY RESIDUE --------------------------------------- GLY A 3 52.00 MSE A 4 62.00 GLY A 5 0.00 ..(略) LEU A 81 56.00 PRO A 82 119.00 <B><FONT COLOR="#FF0000"><!--SUMMARY_BEGIN--> Total area of chain A: 4606.0 -------------------------------------------
この結果は分子Bによってアクセスできなくなった面積が除かれた結果で、4,606Ųと計算されています。
従って、求める接触面積は5,410 - 4,606 = 約800Ųということが分かりました。
もうちょっと簡単に求める
ダイマーぐらいならいいのですが、8merなどになると求めるのが大変です。その場合は、CCP4iからAREAIMOLを使います。
[Structure Analysis] -> [Accessible Surface Areas]で起動します。または[Program List]から[AreaIMol]でも起動できます。
ここで、Run AreaIMol to Calculateの項目を[pairwise area differences between molecules]を選択し、XYZINに対象とするPDBファイルを選択します。使用する分子は後ほど定義しますので、全てのChainおよび水分子が入っていても問題ないようです。
続いて、Molecule Definitionsでそれぞれの分子を定義します。例えばA, B, C, DならMolecule #1,#2,#3,#4を設定します。nameは任意、consists of chainsにChain IDを指定します。
指定が終わったら実行すると全ての組み合わせについて計算してくれます。以下は8merにて実行してみた結果です。全ての組み合わせを計算するので結構時間がかかります(まあ、数分ですが)。
Area excluded on first molecule due to interaction with second ============================================================== A B C D E F G H A - 872.3 319.1 152.3 0.0 0.0 0.0 0.0 B 855.4 - 87.9 324.4 0.0 0.0 0.0 0.0 ... G 0.0 0.0 0.0 0.0 301.7 101.2 - 871.6 H 0.0 0.0 0.0 0.0 105.6 324.1 870.8 - Average areas excluded in pairwise interactions =============================================== A:B 863.85 A:C 321.65 A:D 153.85 A:E 0.0 ...
こんな感じです。
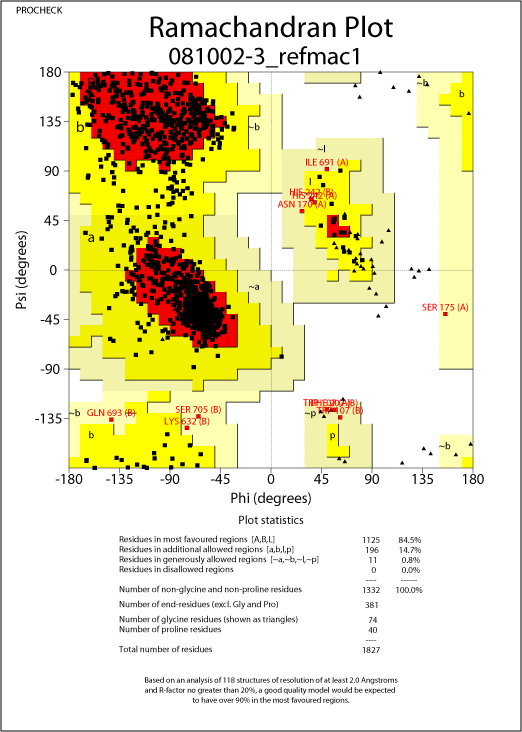
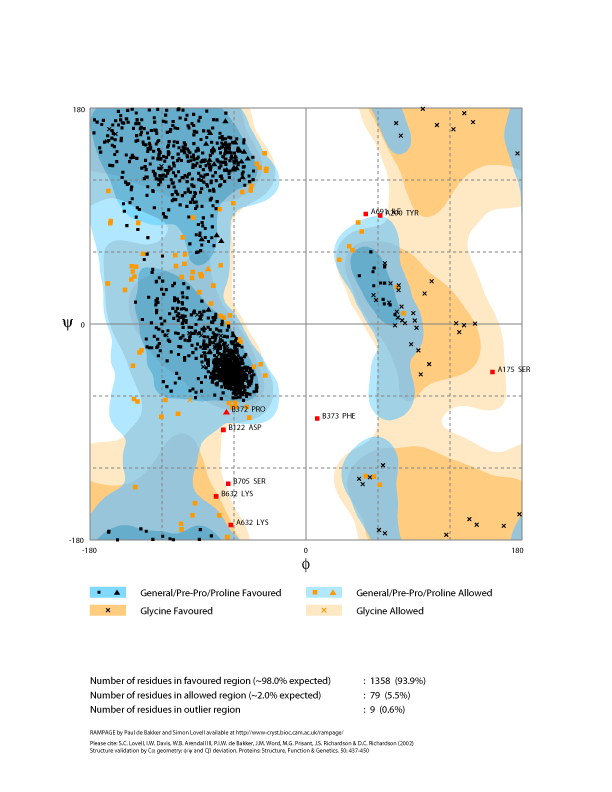
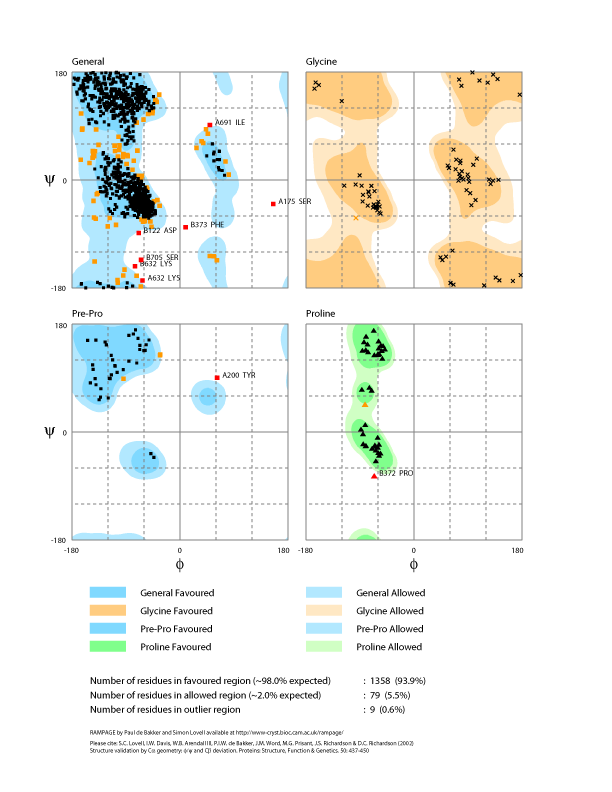
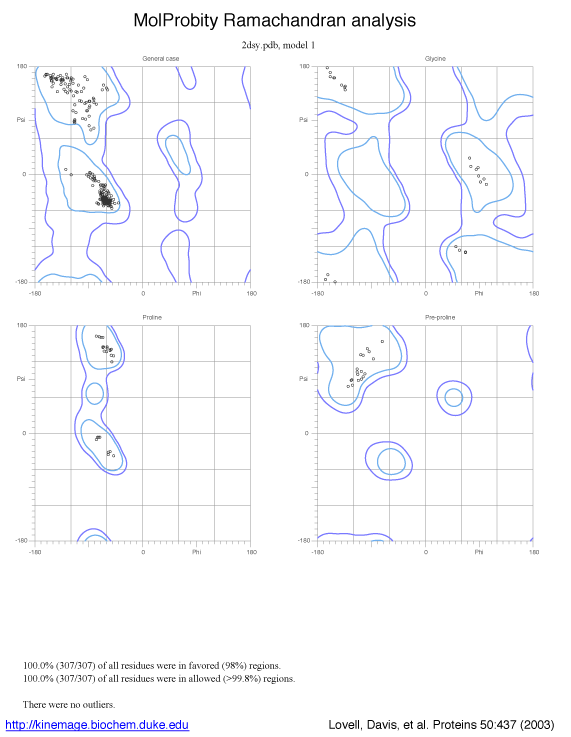
ラマチャンドランプロット(Ramachandran Plot)を描きたい
CCP4のRAMPAGEを使います。
RAMPAGEはRAPPERっていうラマチャンドランプロットを描くプログラムから派生したものだそうで、PROCHECKがかなり古いデータを基にしているのに対して、RAMPAGEは新しいようです。プログラムの説明にはPROCHECKよりもRAMPAGEを使えと書いてあります。論文を投稿した際にもそのように言われることがあるみたいです。
ドキュメントによるとRAMPAGEは、
The Ramachandran diagram plots phi versus psi dihedral angles for each residue in the input pdb file. The diagram is divided into favoured, allowed and disallowed regions, whose contouring is based on density dependent smoothing for 81234 non-Glycine, non-Proline and non-preProline residues with B < 30 from 500 high-resolution protein structures. Regions are also defined for Glycine, Proline and preProline.
ということみたいです。出力もPROCHECKよりおしゃれになってます ![[heart]](image/face/heart.png) 。
。
出力例
出力はPostScript形式ですがPNGにしてあります。クリックで拡大します。
RAMPAGEとMolProbityは計算アルゴリズムが同じようなので同じような結果になるはずです。ただ、ここでは別のタンパクなので出力が異なりますが・・・(^^;。PROCHECKとRAMPAGEは同じタンパクで出力しています。計算結果が異なるのでOuterの数が異なっています。
参考
- CCP4を使ってみよう
- http://www.ccp4.ac.uk/ccp4/html/rampage.html -- CCP4内のプログラムドキュメント
- http://mordred.bioc.cam.ac.uk/~rapper/rampage.php -- RAMPAGE計算サーバ
- http://molprobity.biochem.duke.edu/ -- MolProbity(構造チェックサーバ)
- S.C. Lovell, I.W. Davis, W.B. Arendall III, P.I.W. de Bakker, J.M. Word, M.G. Prisant, J.S. Richardson, D.C. Richardson, Structure validation by Calpha geometry: phi,psi and Cbeta deviation. Proteins: Struct. Funct. Genet. 50 437-450 (2003).
モデルとして置いた化合物を精密化したい
CNSのmodel_anneal.inpなどを使います。
化合物の結合様式をモデルで示したい場合、実験データがないので通常の精密化は行えません。その時はCNSでMDっぽいことができるのでそれを行ってみます。Rigid body, Annealing/Molecular Dynamics, Energy minimaizationが行えます。
二次構造をアサインしたい
2010-03-01
DSSPやSTRIDEなどを使います。
どういった基準で二次構造(つまりへリックスやストランド)を割り当てるかはパラメータにも依存すると思うので一概には言えないと思いますが、標準的なものを使うならDSSP、またはどの程度使われているかは知らないのですがさっき見つけた ![[huh]](image/face/huh.png) STRIDEを使います。
STRIDEを使います。
DSSPは非常に細かい二次構造パラメータを出力してくれます実力派、STRIDEはWebインタフェースも用意されており、テキスト出力や図による出力に対応しています。結果の違いは比べてないのですが、二次構造を割り当てた場合は使用したプログラムを明記した方がいいでしょう。このあたりの結果は、PyMOLなどで描かせた割り当てとは異なるのでいつも扱いに苦慮します・・・
電子密度図
精密化済みの構造からオミットマップを描きたい
2010-04-28
REFMAC+FFTまたはCNSを使います。
すでに精密化済みの構造を基にしてオミットマップを描く方法はCCP4を使う方法およびCNSを使う方法が考えられます。私が思いついた方法では共にコマンドスクリプトで行うので慣れているプログラムを使用して下さい。個人的な感覚では、CCP4の方が手間が少ないように感じました。
残基をオミットしたPDBファイルを用意し、CCP4またはCNSを使用してマップを描きます。
- CCP4で行う
CCP4パッケージのREFMACおよびFFTを使用します。COOTで位相情報のないMTZを用いてマップを描かせると使われる方法です。手順は以下の通り。- REFMACで精密化せずに位相情報付きのMTZを作成する
- FFTでマップを描く
この作業を行うためのスクリプトは以下になります。
-
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15
#!/bin/sh refmac5 XYZIN omit.pdb HKLOUT tmp.pdb HKLIN start.mtz HKLOUT phased.mtz <<EOS MAKE HYDROGENS NO NCYCLES 0 WEIGHT AUTO LABIN FP=F SIGFP=SIGF EOS rm -f tmp.pdb fft HKLIN phased.mtz MAPOUT fofc.map <<EOS SCALE F1 1.0 LABIN F1=DELFWT PHI=PHDELWT W=FOM GRID SAMPLE 3 EOS
FFTコマンド中のGRID SAMPLE 3の値を大きくするとマップのメッシュが細かくなり、キレイなマップが描けます。ディフォルトは3です。 -
- CNSで行う
CNSでも同様の作業を行うことができます。使用するインプットファイルはmodel_map.inpです。
- 参考:CNSを使ってみよう